
Nucleic Acids Research|一种用于新月柄杆菌高效快速基因组编辑的CRISPR/SpCas9M报告系统
新月柄杆菌(Caulobacter crescentus)因其独特的不对称分裂特征常被用于研究细胞分化、极性分裂以及与细胞周期相关的分子调控机制,被认为是研究单细胞向多细胞生命转变的模式微生物。然而,新月柄杆菌的遗传操作手段费时、费力且效率低,严重影响了对其深入研究的潜能。
近日,中国科学院深圳先进技术研究院合成生物学研究所赵国屏院士,赵维研究员团队联合上海交通大学在Nucleic Acids Research上发表题为“A CRISPR/SpCas9M-reporting system for efficient and rapid genome editing in Caulobacter crescentus”的方法论文,针对上述问题,开发了一套基于CRISPR/SpCas9M的基因编辑报告系统,实现了对新月柄杆菌高效、快速且无痕的基因编辑。该项工作不仅为新月柄杆菌的遗传操作提供了强有力的工具,也为解决其它难以进行遗传操作的非模式菌提供了新的技术思路和方法学参考。
设计:CRISPR/SpCas9M-报告系统
研究团队最初构建了一个基于同源重组(HR)的CRISPR/Cas系统。将Streptococcus pyogenes来源的SpCas9、sgRNA和同源臂(H-arms)克隆至携带pBBR1复制子的复制型质粒pBXMCS2中。为了实现无痕编辑,在同源臂之间未设计任何抗生素抗性基因。然而,将该编辑质粒电转化至新月柄杆菌后,发现获得极少甚至无菌落,并且存活菌株的靶位点均未发生预期编辑。这表明CRISPR/SpCas9的切割具有致死性,而该菌细胞内的同源重组(HR)效率又相对较低。
为实现有效的靶向基因编辑,研究团队对编辑质粒进行了系统性分析,并围绕三个关键方向进行了优化。
首先,研究人员筛选了来自不同物种的Cas蛋白,包括Streptococcus pyogenes Cas9 (SpCas9)、Francisella novicida Cas12a (FnCas12a), Streptococcus thermophilus CRISPR1-Cas9 (Sth1Cas9)和Streptococcus thermophilus CRISPR3-Cas9 (Sth3Cas9),以评估它们在新月柄杆菌中的编辑效率,结果发现上述所有Cas蛋白均未检测到编辑克隆。然而,当根据新月柄杆菌的密码子偏好性优化SpCas9编码序列(命名为SpCas9M)后,基因编辑效率可达到15%左右。
其次,研究团队通过调控SpCas9M的表达水平来提升编辑效率。研究人员测试了两种诱导型启动子,香草酸诱导型启动子(Pvan)和木糖诱导型启动子(Pxyl),检测不同诱导剂浓度下的基因编辑效率。结果发现相对较低的SpCas9M表达水平更有利于新月柄杆菌的基因组编辑,编辑效率最高可达到40%左右。
最后,通过分析未发生基因组编辑的克隆中的编辑质粒,研究人员惊奇的发现这些克隆的SpCas9M编码序列均存在缺失。这一现象证实了CRISPR/SpCas9系统的致死性,并表明新月柄杆菌中存在着强烈的选择性压力。基于此,研究人员提出假设:通过预先识别并排除这些SpCas9M突变体,可有效提升在新月柄杆菌中的表观编辑效率。为此,研究人员在SpCas9M的C端融合了超折叠绿色荧光蛋白(sfGFP)作为报告系统,通过荧光信号指示,在菌落PCR前筛选并排除SpCas9M异常表达的克隆。最后,总体实现编辑效率达到80%左右,研究团队将其命名为CRISPR/SpCas9M-报告系统。
应用1:基因的单敲除、双敲除和敲入
在新月柄杆菌中,不对称细胞分裂由一对激酶(DivJ)和磷酸酶(PleC)协同调控。DivJ与PleC在细胞两极的差异性定位形成磷酸化梯度,从而调控子代细胞的命运决定。值得注意的是,DivJ和PleC的极性定位并非主动过程,而是通过不同脚手架蛋白介导的招募机制实现。已有研究表明,位于细胞旧极的DivJ由PopZ-SpmX脚手架蛋白复合体招募,而位于细胞新极的PleC则通过脚手架蛋白PodJ实现定位。
为在遗传学层面验证上述蛋白互作关系,研究人员利用CRISPR/SpCas9M-报告系统对新月柄杆菌中的spmX和podJ基因分别进行了敲除。随后,研究人员将mCherry荧光标记的DivJ和PleC蛋白分别转化至上述敲除菌株中,并利用倒置荧光显微镜观察其定位情况。通过柄状结构指示细胞极性发现:在野生型菌株中,DivJ-mCherry特异性富集于细胞旧极,而PleC-mCherry则特异性定位于细胞新极;然而,当spmX基因被敲除后,DivJ-mCherry丧失极性定位能力;类似地,敲除podJ基因后,PleC-mCherry在细胞内呈现弥散性分布。上述结果表明,脚手架蛋白SpmX和PodJ通过直接相互作用维持DivJ/PleC的极性定位。
研究人员还依次敲除podJ和spmX基因构建了双突变菌株,双突变株ΔpodJ-ΔspmX中DivJ-mCherry与PleC-mCherry均呈现弥散分布。值得注意的是,敲除podJ不影响DivJ-mCherry的定位,敲除spmX也不影响PleC-mCherry的定位,表明脚手架蛋白与信号蛋白间存在正交调控关系。
迄今为止,科学家尚未能实现新月柄杆菌中无标记的基因插入。为解决这一难题,研究人员应用CRISPR/SpCas9M-报告系统测试了靶向基因插入的可行性。利用基因敲入进行功能获得性研究时,研究人员选择中性插入位点(Neutral Insertion Site, NIS)以避免基因极性效应。研究人员通过targetFinder鉴定出一组新月柄杆菌潜在中性插入位点,并选取评分最高的NIS位点。当利用CRISPR/SpCas9M-报告系统将Pcat-mcherry敲入到中性位点后,倒置荧光显微镜下可观察到菌株内的mCherry红色荧光信号,证实该基因被正确插入且有效转录翻译。此外,敲入菌株均未检测到明显的生长及形态学改变,表明这些插入位点不会对新月柄杆菌的适应性造成显著影响。
应用2:在中华根瘤菌和农杆菌中应用CRISPR/SpCas9M-报告系统
作为新月柄杆菌的近缘物种,中华根瘤菌(S. meliloti)与农杆菌(A. fabrum)分别是植物共生菌与病原菌,二者在现代农业生物技术中均具有重要价值。其中,苜蓿中华根瘤菌近年来还被用作天然产物发酵的细胞工厂。然而,由于缺乏有效的细胞同源重组活性,这两种细菌均面临高效基因编辑的难题。近来,针对农杆菌与中华根瘤菌已分别开发出基于CRISPR的碱基编辑器与转座子介导的基因组编辑工具。尽管如此,对于经典的基因敲除与插入仍是该领域面临的重要技术挑战。
为此,研究人员测试了CRISPR/SpCas9M-报告系统在农杆菌与中华根瘤菌中的编辑能力。实验选择编码胸苷激酶的tdk基因作为靶标,该酶通过将5-氟-2′-脱氧尿苷(5-FUdR)磷酸化为毒性类似物氟-dUMP(F-dUMP),使细胞对5-FUdR敏感。为验证编辑菌株表型,在含200 μg/ml 5-FUdR的PYE平板上进行生长实验。结果显示:Δtdk菌株可在含5-FUdR的平板上存活,而野生型中华根瘤菌与农杆菌则无法生长。
综上所述,本研究开发的CRISPR/SpCas9M-报告系统相较于现有工具展现出显著优势,其成功应用于中华根瘤菌与农杆菌等农业重要微生物的基因组编辑,表明该系统可进一步扩展至微生物组研究或生物制造领域相关的其他遗传操作困难物种。此外,该系统支持快速、迭代的遗传操作,为基因功能研究与合成通路构建提供了理想工具。未来,对CRISPR逃逸机制的深入解析将指导设计更为稳健的CRISPR/Cas工具。CRISPR/SpCas9M-报告系统的建立不仅彰显了可靠基因组编辑平台的重要性,更为微生物遗传学、合成生物学及其他生物技术领域的创新发展提供了基础。
中国科学院深圳先进技术研究院研究员赵维、上海交通大学教授艾连中为本文共同通讯作者。中国科学院深圳先进技术研究院助理研究员孙敬贤、研究助理余昕为共同第一作者。本工作获得了中国科学院院士赵国屏、中国科学院深圳先进技术研究院研究员戴磊和山东大学教授王海龙的大力支持和帮助;本研究获得了国家重点研发计划、中国科学院战略性先导科技专项、国家自然科学基金、广东省基础与应用基础研究基金,以及深圳合成生物学创新研究院科研计划等多个项目的资助。
文章上线截图
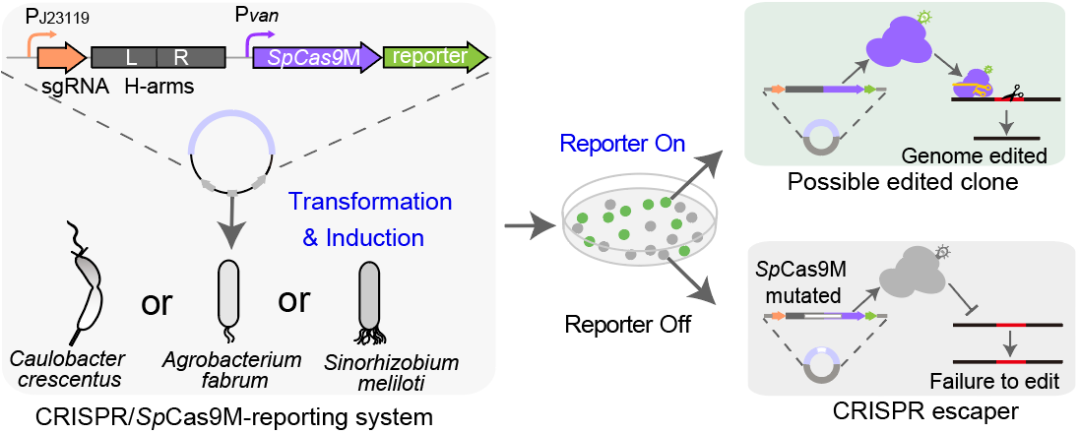
图1. CRISPR/SpCas9M-报告系统的作用原理
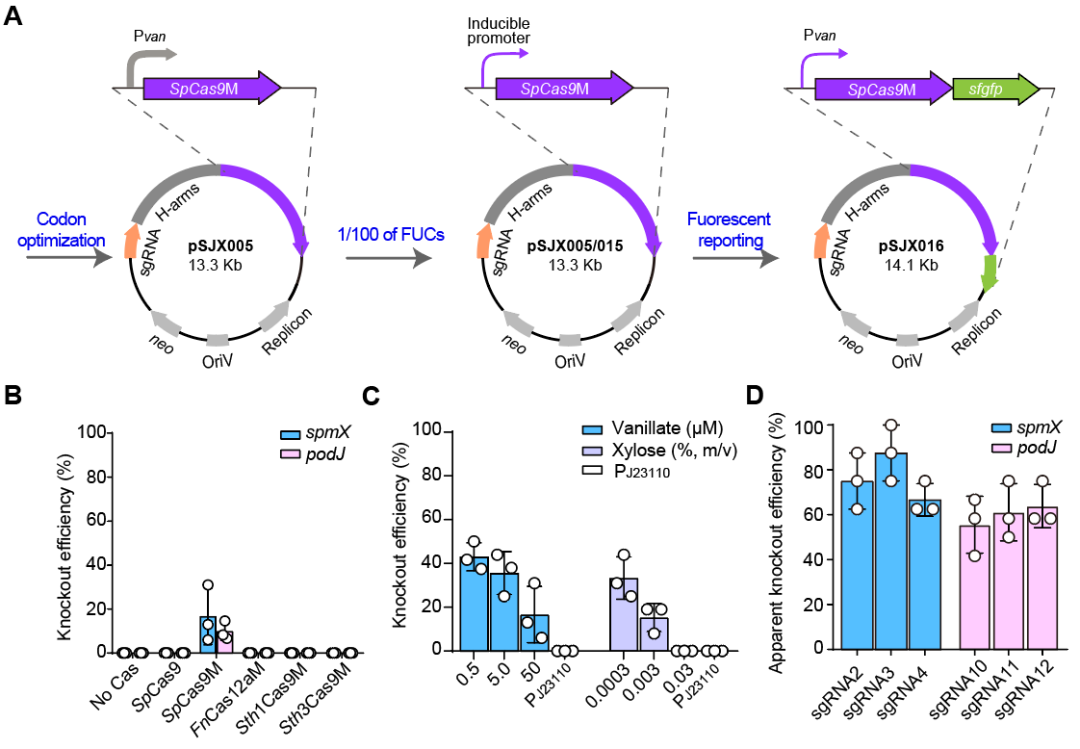
图2. CRISPR/SpCas9M-报告系统的搭建过程
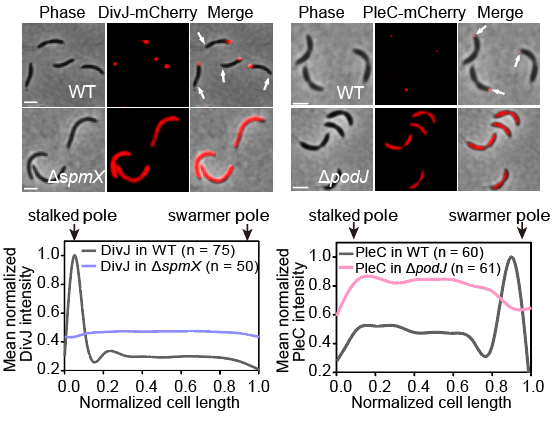
图3. DivJ-mCherry和PleC-mCherry分别在单敲除突变体ΔpodJ和ΔspmX中的分布

图4. DivJ-mCherry和PleC-mCherry在双敲除突变体ΔpodJ-ΔspmX中的分布
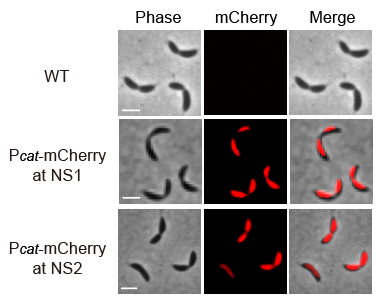
图5. 在NIS位点敲入Pcat-mcherry后的表型鉴定
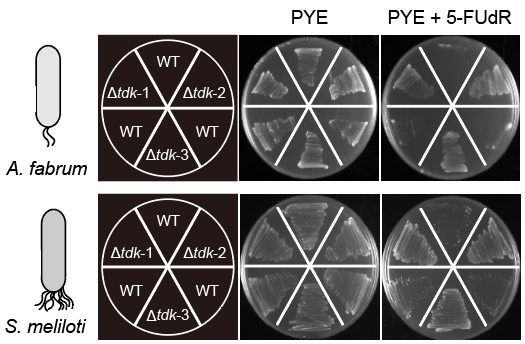
图6. tdk敲除突变体的鉴定
附件下载: