
Nucleic Acids Research | 基于深度学习与绝缘化原理的合成生物顺式调控元件从头设计
在合成生物学领域,科学家们希望能“编程”生命,实现可预测地设计基因元件(比如启动子、增强子)、蛋白质等目标,让细胞按照人们预定的强度表达功能基因。近年来,人工智能(AI)特别是深度学习技术,成为这项工作的“新引擎”。通过分析实验数据,AI模型能预测哪些序列会带来强或弱的基因表达,甚至能设计出全新的调控序列。
然而,这项技术存在一个长期被低估的难题——“数据污染”。正如人们所讨论的,大语言模型会受到网络中“错误信息”的污染,其本质在于训练数据受到非目标信息干扰,导致模型学习到错误的规律。在常规生物实验中,研究者会在特定宿主细胞中对人工设计的序列进行测试。但很多看起来“活跃”的序列,其活性实际上源于宿主细胞自身的意外激活,而非目标元件本身的活性。把这类“污染”数据喂给AI模型,就如同教幼儿识字时混进错别字,AI模型也会因此“学偏”,记住不应有的规则。这不仅会导致模型的预测结果失真,还使其难以在不同物种间实现通用。
近日,中国科学院深圳先进技术研究院定量合成生物学全国重点实验室、合成生物学研究所娄春波课题组与清华大学自动化系汪小我课题组合作开展的研究取得重要进展。他们提出并验证了一项关键观点:去除宿主细胞内“污染语料”,是实现高精度模型预测及可控从头设计顺式基因元件的前提条件。相关成果以“De novo design of insulated cis-regulatory elements based on deep learning-predicted fitness landscape”为题发表在国际学术期刊Nucleic Acids Research上。
问题发现:数据污染是模型“预测失灵”的根源
在利用深度学习设计基因调控元件的过程中,存在一个常被忽视但至关重要的问题——宿主背景污染。研究团队在分析K1.5启动子系统的实验数据时发现:当采用随机序列筛选活性启动子时,许多看似“活跃”的序列,其活跃并非源于对目标RNA聚合酶(K1.5 RNAP)的调控,而是因意外被宿主大肠杆菌自身的转录体系激活所致。
这类似于教AI识别苹果图片时,训练数据中混入了橘子图片却都标注为 “苹果”;在此情况下,AI模型学到的不是真正区分苹果的特征,而是各种混杂的错误模式。
研究人员通过深入分析发现,这种“宿主背景污染”并非个别现象,而是在宿主细胞中任意筛选随机序列时普遍存在的问题。在传统体系里,顺式调控元件必须和宿主的转录因子(比如RNA聚合酶、σ因子等)协同作用,因此随机序列极易无意中激活宿主自身的调控机制,产生“伪阳性”信号。
这类“非目标”信号会对AI模型形成误导,使得它学到的规律仅在特定宿主内有效,无法迁移到其他物种或新的表达系统。要真正实现可预测、可迁移的功能元件设计,就必须从源头上去掉此类背景干扰,建立一个真正“正交”(即彼此独立、互不干扰)的表达系统,确保AI模型学到的调控规律具有纯粹性、可解释性和可泛化性。
为此,研究团队设计了一套“预测+实验双重筛选”的数据净化流程:首先通过模型预测识别并排除可能受宿主背景激活的序列,再借助双通道诱导实验(有/无IPTG条件)进一步筛掉对目标RNA聚合酶无响应的序列。最终,团队构建出一个仅包含K1.5系统真实调控信息、宿主背景干扰最小化的高质量数据集。
建模突破:构建绝缘表达系统,绘制真实的全景观活性功能
基于上述净化后的高质量数据集,研究团队训练了一个深度卷积神经网络模型。模型以DNA启动子序列的编码作为输入,以实验测得的表达强度作为输出。
不同于传统仅能给出结果预测的“黑箱”模型,团队通过特征可视化分析,成功绘制出“活性功能全景观”。这一“景观”可类比表达强度随DNA序列变化的地形图。模型能在该景观里找到“局部高峰”(即表达强度最优的序列模式),还能识别出关键的功能motif(序列片段),从而帮助解析基因调控的内在规律。
一个极具意义的发现是:仅需大约1250条经净化的高质量序列,即可把模型的表达强度预测精度做到R²=0.90。这表明数据的“纯净度”比规模更重要。该结果为后续利用生成模型设计新序列打下了坚实的基础。
在这一精准的表达景观模型基础上,团队开发出真正的“从头设计(de novo design)”策略。从完全随机生成的DNA序列出发,利用模型预测到的“爬坡”方向,通过反向传播和迭代优化,持续调整碱基组成,让序列在“表达景观”中逐步攀升至目标表达强度区域。
这一方法突破了以往以来天然模板、通过反复突变和筛选实现的“半理性设计”模式,实现了真正意义上的“从零生成”。实验验证显示,该方法设计出的人工启动子其表达强度范围广泛覆盖野生型水平,且预测值和实际测试结果高度一致,尤其在中高表达区的偏差极小,且设计出的不同序列之间差异显著(Hamming距离大于10bp),有效规避了同源重组或序列冗余问题,保证了多样性和稳定性。
功能验证:生成启动子在不同宿主中保持表达可预测性
为进一步验证所设计调控元件的功能稳定性与跨物种适应性,研究团队将部分模型生成的启动子序列移植至哺乳动物细胞系统中进行表达测试。实验选取常用的中国仓鼠卵巢细胞(CHO)为代表,在等效的启动子-RNAP组合条件下评估其表达活性。
结果显示,这些已在大肠杆菌中验证的人工启动子,在CHO细胞中同样呈现出与模型预测值基本一致的表达趋势,其表达强度与模型预测结果间具有显著线性相关性(R² = 0.54)。尽管不同物种的表达背景存在差异,该结果仍表明,模型设计出的顺式元件具备良好的表达可控性和宿主迁移能力,具备“跨宿主平台”通用化应用的潜力。
为评估该策略的系统适配性,研究团队进一步将活性功能景观建模与从头设计方法拓展至T7 RNA聚合酶系统。作为经典的合成表达平台,T7系统具有较强的表达能力和广泛的应用基础。研究显示,所生成的T7启动子序列同样实现了表达水平的可控设计,且与模型预测结果高度一致。这一结果验证了该方法不仅适用于K1.5系统,还具备向其他单因子驱动、正交表达系统泛化的能力,为调控元件的模块化设计与系统工程化打下了通用基础。
本研究建立了一套面向顺式调控元件的高通量、可解释、跨系统泛化的从头设计流程,有望解决以往因为宿主背景干扰导致的模型预测失真和迁移失败这一长期难题。通过结合绝缘型表征系统的构建和深度学习预测模型,研究团队实现了从随机序列到目标功能启动子的精准生成,并验证了其在不同RNA聚合酶系统和不同宿主细胞中的通用性。该成果为合成生物线路设计、跨物种基因回路构建以及可编程细胞工厂的开发提供了全新解决方案,或将推动AI驱动的基因调控研究迈向真正的“功能级别智能设计”阶段。
中国科学院深圳先进技术研究院研究员娄春波、清华大学教授汪小我为本文共同通讯作者。清华大学博士研究生王昊晨,中国科学院深圳先进技术研究院助理研究员项延会、研究助理刘子明为共同第一作者。研究工作得到了国家重点研发计划、国家自然科学基金、中国科学院青年交叉科学团队项目以及深圳合成生物学创新研究院等项目的联合资助。

文章上线截图
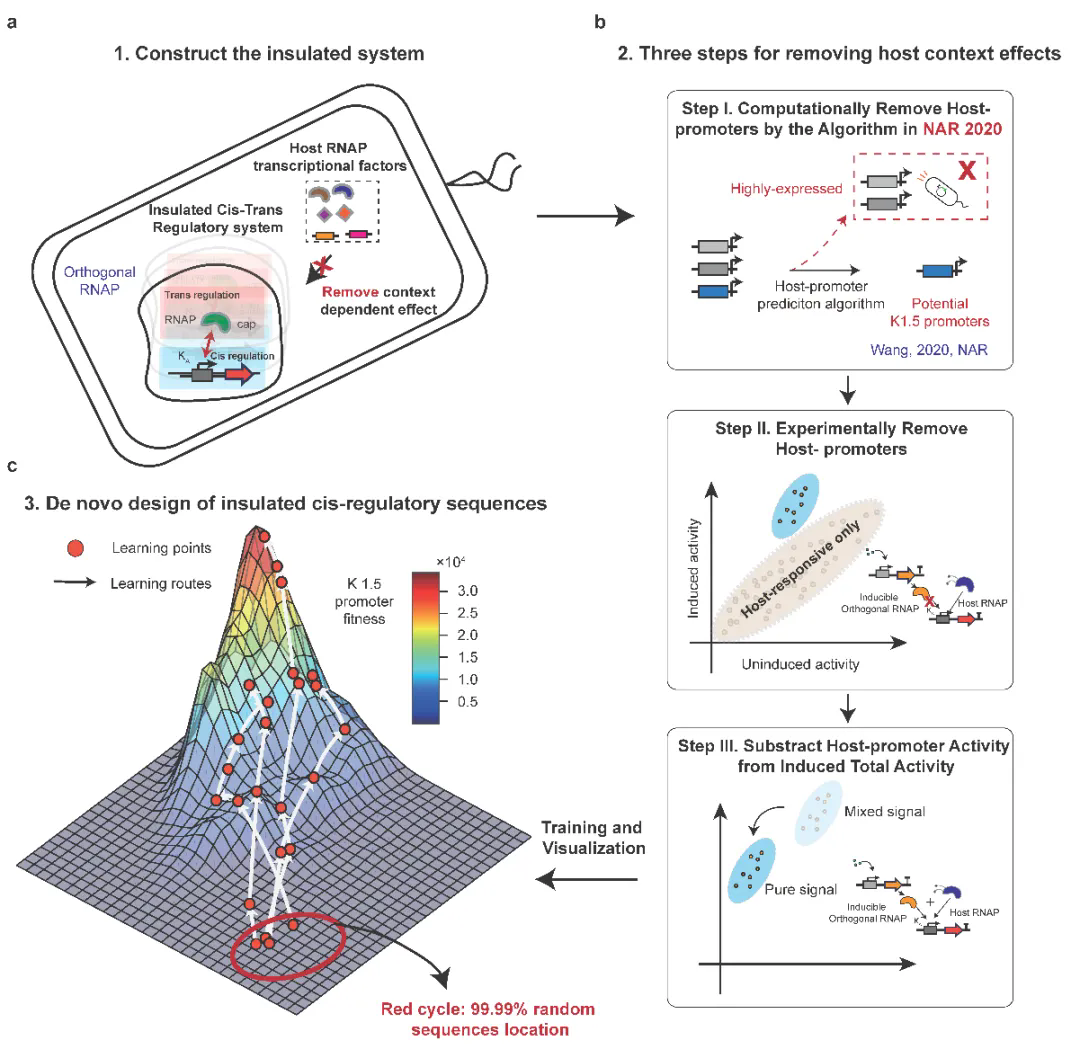
图1 研究人员构建的正交调控系统示意图
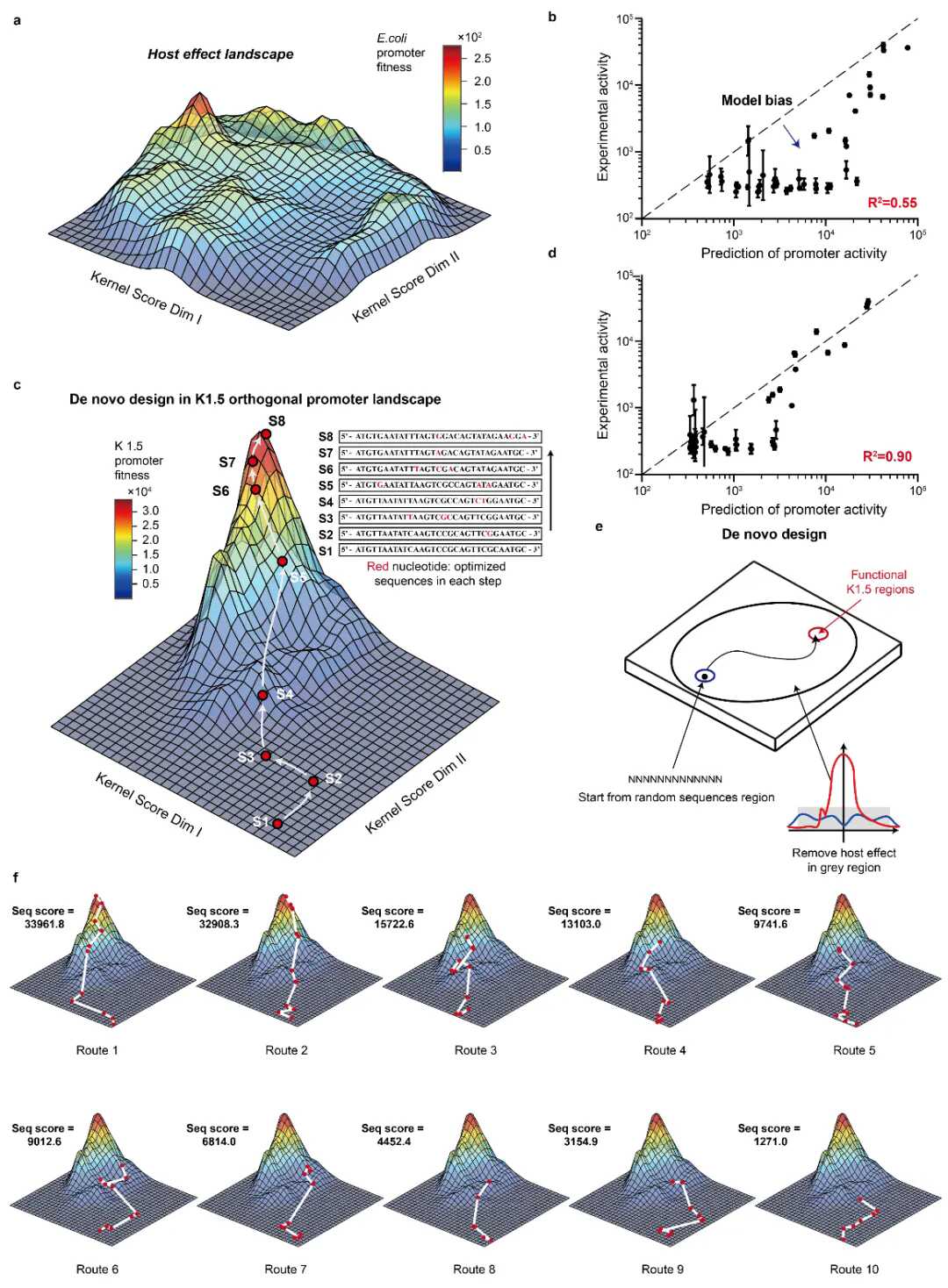
图2 从头设计具有功能与序列多样性的调控元件序列

图3 从头设计的绝缘型启动子在哺乳动物细胞中的泛化评估及其在T7正交系统中的设计策略拓展
附件下载: